- * 프린트는 Chrome에 최적화 되어있습니다. print
저희 연구실은 DNA 손상 반응 경로와 만성 골수성 백혈병(CML)의 발명/진행을 중점적으로 연구하고 있습니다. DNA 손상 반응은 진핵 세포에서 유전적 안정성을 유지하는 데 중요합니다. ATM, ATR, MDC1, RNF8, RNF168, RAP80, BRCA1 및 BRCA2를 포함한 많은 종양 억제 단백질이 DNA 손상경로에 관여하기 때문에 이 경로에 대한 완전한 이해는 종양 발생을 예방하는 데 중요합니다. 따라서 우리 실험실의 목표는 DNA 손상 신호 경로를 연구하여 종양 발생의 유전적 안정성과 분자 메커니즘을 이해하는 것입니다. CML 연구는 저희 연구실의 두 번째 프로젝트입니다. 저희 연구실은 CML의 티로신 키나제 억제제에 내성이 있는 유전자를 식별하는 데 중점을 두었습니다. 또한 발견된 유전자의 분자 메커니즘을 연구하여 CML 환자에게 적용하고 있습니다. 이 연구 프로젝트의 궁극적 목적은 만성 골수성 백혈병의 치료 및 진단에 기여하는 것입니다.
My lab focuses on studying the DNA damage response pathway and development/progression of chronic myeloid leukemia (CML). DNA damage responses are important in maintaining genetic stability in eukaryotic cells. Because many tumor suppressor proteins including ATM, ATR, MDC1, RNF8, RNF168, RAP80, BRCA1, and BRCA2 are involved in the DNA damage pathway, the complete understanding of this pathway is important in preventing neoplastic transformation. So, our laboratory’s goal is to understand the inherent genetic stability and molecular mechanisms of tumorigenesis by studying DNA damage signaling pathways. CML study is the second project in my Lab. My Lab has focused on identifying genes that are resistant to the tyrosine kinase inhibitor of CML. In addition, we are studying the molecular mechanisms of the discovered genes and applying them to CML patients. The aim of this research project is to ultimately contribute to the treatment and diagnosis of chronic myelogenous leukemia.
Major research field
DNA damage, Cancer, CML
Desired field of research
Research Keywords and Topics
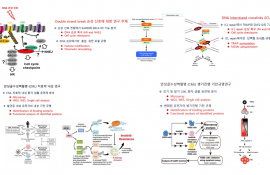
1. DNA 손상 연구
(1)Double strand break 손상 신호에 대한 연구 주제
1) 손상 신호 전달에서 RAP80와 BRCA1의 기능 분석
- DNA 손상 복구 (HR and NHEJ)
- Cell cycle checkpoint
2) DNA 손상에서의 후성 유전 조절 규명
- Histone modification
- Chromatin remodeling
(1) DNA Interstrand crosslinks (ICLs) repair 연구
1) ICL repair에서 TRAIP와 ZNF212의 기능 분석
- ICL repair 손상 복구 (FA and NEIL3 pathway)
- Cell cycle checkpoint
2. 백혈병 진행 연구
(1) CML 치료제 내성 및 병기 진행 환자 샘플 유전체 분석
- Microarray
- WGS, WES, Single cell analysis
(2) CML 치료제 내성 및 병기 진행 기전 분석
- Identification of binding proteins
- Functional analysis of identified proteins
Research Publications
MORE• Nucleic Acids Res. Alteration of replication protein A binding mode on single-stranded DNA by NSMF potentiates RPA phosphorylation by ATR kinase. Kang Y, Han YG, Khim KW, Choi WG, Ju MK, Park K, Shin KJ, Chae YC, Choi JH, Kim H, Lee JY. 2023
• Nucleic Acids Res. ZNF212 promotes genomic integrity through direct interaction with TRAIP. Chung HJ, Lee JR, Kim TM, Kim S, Park K, Kim MJ, Jung E, Kim S, Lee EA, Ra JS, Hwang S, Lee JY, Schärer OD, Kim Y, Myung K, Kim H. 2023
• Leukemia. Cobll1 is linked to drug resistance and blastic transformation in chronic myeloid leukemia. Han SH, Kim SH, Kim HJ, Lee Y, Choi SY, Park G, Kim DH, Lee A, Kim J, Choi JM, Kim Y, Myung K, Kim H, Kim DW. 2017
• Nature communication. TRAIP/RNF206 is required for recruitment of RAP80 to sites of DNA damage. Lee NS, Chung HJ, Kim HJ, Lee SY, JJi J, Seo Y, Han SH, Choi M, Yun M, Lee SG, Myung K, Kim Y, Kang HC, Kim H. 2016
국가과학기술표준분류
- LA. 생명과학
- LA01. 분자세포생물학
- LA0101. 신호전달




